轴手性联芳基化合物的高效生物合成平台
2025年9月10日,北京生命科学研究所 / 清华大学生物医学交叉研究院刘臻实验室与黄牛团队合作,在Nature Synthesis期刊发表了题为“Molecular-dynamics-simulation-guided directed evolution of flavoenzymes for atroposelective desaturation”的研究论文。该研究报道了一种基于定向进化策略开发的创新性生物催化方法,研究团队利用来源于Parageobacillus thermantarcticus的黄素酶PtOYE,通过工程化改造获得了具有对映选择性去饱和活性的变体,从而实现了轴手性联芳基化合物的高效合成。该项目主要内容由刘臻实验室2022级TIMBR项目的博士研究生尹红宁和黄牛实验室的博士研究生陈钊(现为博士后)完成。刘臻研究员和黄牛研究员为论文通讯作者。
轴手性(Atropisomerism)是一类因单键旋转受限而形成的稳定立体异构体。与传统的手性中心不同,轴手性分子依靠空间取向的差异实现立体选择性,其构型稳定性主要取决于旋转势垒及空间位阻效应。这类化合物广泛存在于天然产物、药物分子以及手性配体和有机小分子催化剂中,具有重要的化学和生物学意义。因此,如何高效且选择性地构建轴手性化合物,一直是合成化学和生物催化领域的研究热点。
在生物催化领域,轴手性化合物的对映选择性合成主要有两种策略。第一种基于已有的(潜)手性轴,通过天然酶(如脂肪酶lipase、酮还原酶KREDs、亚胺还原酶IREDs等)催化的不对称转化(如动力学拆分)来实现手性轴的选择性构建。第二种方法则是直接构建新的手性轴,例如由漆酶或细胞色素P450催化的酚类化合物氧化偶联反应。此外,也有报道利用钯依赖的人工金属酶通过Suzuki偶联反应制备轴手性联芳基化合物。尽管相关研究已取得重要进展,但总体而言,生物催化在轴手性骨架的构建方面仍局限于少数的逆合成策略和反应类型,与化学合成方法相比仍存在较大差距。
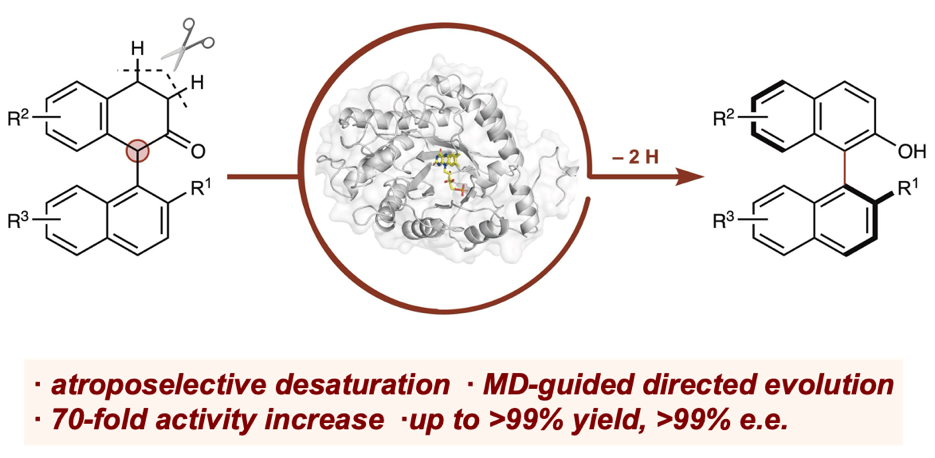
近日,北京生命科学研究所 / 清华大学生物医学交叉研究院刘臻实验室与黄牛团队合作,报道了一种基于定向进化策略开发的创新性生物催化方法,研究团队利用来源于Parageobacillus thermantarcticus的黄素酶PtOYE,通过工程化改造获得了具有对映选择性去饱和活性的变体,从而实现了轴手性联芳基化合物的高效合成。值得一提的是,分子动力学模拟(MD simulation)在定向进化进程中发挥了关键作用,该技术揭示了催化反应过程中酶的潜在构象变化,并在此基础上识别出此前在PtOYE及其同类酶中未被发现的重要残基。以此为指导获得的工程化酶ADes-5相较于初始变体(PtOYE-Y28F),其催化效率(kcat/KM)提升了70倍以上。此外,研究团队通过X射线晶体学手段解析了ADes-5的结构(PDB:9IUV),确定了其活性中心和整体的结构特征。该工程化酶能够将多种1-芳基-2-四氢萘酮底物以很高的收率和良好的对映选择性转化为相应的联芳基产物,进一步的衍生反应展示了合成产物在多官能团轴手性分子构建中的广泛应用潜力。研究组不仅展示了酶催化脱氢反应在轴手性选择性合成中的潜力,也突显了黄素酶在合成轴手性分子方面的重要能力,对有机合成与药物发现都具有重大意义。同时,该研究也验证了MD在预测蛋白质潜在构象变化并揭示蛋白功能的结构机制中的准确性和重要作用。
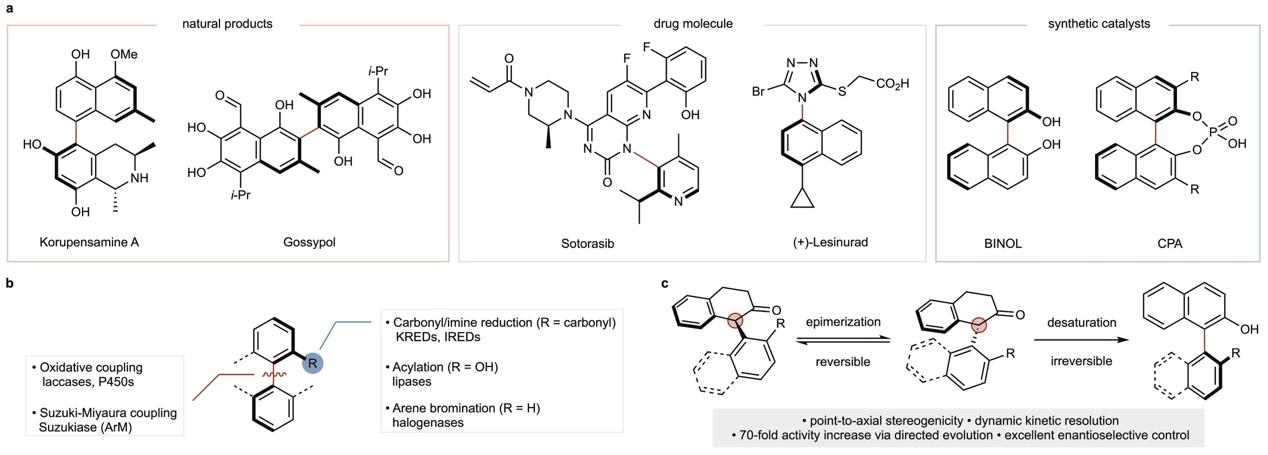
图1:轴手性化合物的生物催化策略。
分子动力学模拟指导的蛋白质工程
实验人员从底物1a的动态动力学拆分为切入点,开展了酶催化轴手性脱氢反应的系统研究。首先,作者用实验室自有酶库中的黄素酶对1a的去饱和反应性进行了评估,其中活性最高的是PtOYE-Y28F (ADes-1),对映选择性为82%,催化产率仅为2.2%。为提升ADes-1的催化性能,研究团队随后针对已知影响PtOYE及其同源酶多种酶促反应效率的八个位点进行了位点饱和突变。尽管进行了大量筛选,仅发现一个有益突变A104G,其对产率和对映选择性的改善非常有限。
为提高筛选效率,作者采用了MD模拟指导的蛋白质工程策略。作者首先基于AlphaFold2预测的ADes-1结构以及PtOYE的催化机制,将底物1a的两种对映体分别对接至酶的活性口袋中。其中R构型由于在对接过程中与活性位点存在严重空间位阻而被排除;而 (S)-1a存在两种可能的结合模式,这两种结合模式的主要差异存在于萘环取向:在Conf-I模式中,底物甲氧基朝向活性位点外侧;Conf-II模式中,底物甲氧基朝向活性位点内侧并与S249和R215形成相互作用(图2b)。
作者在此基础上对两种结合模式的复合物结构分别进行了MD模拟,模拟结果显示在Conf-II模式中,随着底物分子的结合,PtOYE蛋白发生了显著的构象变化:Loop I和Loop II之间的“锁扣”结构打开,同时Loop I向内关闭稳定了底物结合(图2d);而在Conf-I模式的MD结果中,未观察到类似的构象变化,同时底物分子结合不稳定(图2c)。MD模拟说明了Conf-II模式的合理性以及构象变化在稳定底物结合过程中的重要性,揭示了PtOYE催化底物反应的潜在结构动态机制。
对模拟结果中的结构进一步可视化分析表明:(1) 在初始构象中,Loop II上的F124与Loop I上的P255和A252通过疏水相互作用形成“锁扣”结构,限制了Loop I的闭合;(2) 在最终的Loop I“闭合”构象中,疏水残基V254靠近底物并形成新的相互作用(图2d)。基于这些结构与动态信息,作者对F124、V254和P255三个位点进行了位点饱和突变筛选,旨在打破“锁扣”构象并稳定“闭合”构象,从而促进底物的结合和催化。筛选得到的F124Q与V254F变体分别对反应收率提升了3.8倍和3.4倍,二者组合后得到的双突变体ADes-4,使产率提升至50%,对映选择性达到88% e.e.。进一步在A252位点进行突变和筛选,获得了催化性能最优的ADes-5,其产率高达98%,对映选择性为89% e.e.,和初始变体ADes-1相比,产率提升了45倍。

图2:分子动力学模拟指导的定向进化。
酶动力学参数的测定
为深入理解蛋白质工程对于轴手性脱氢酶性能的影响,作者测定了进化谱系中四个ADes变体的动力学参数。实验结果表明,F124Q和V254F两个突变显著提高了酶的催化活性,使ADes-4的催化速率(kcat)和ADes-1相比提高了26倍,同时表现出更低的KM值,表明底物亲和力增强。最终获得的ADes-5变体展现出最优的催化性能,其催化效率kcat/KM较初始变体提高了70倍。这些观察结果与作者基于MD模拟所建立的机制模型相一致。具体而言,当F124和A252突变为亲水残基谷氨酰胺时,“锁扣”构象中Loop I和Loop II之间的疏水相互作用被削弱,加速了这两个loop的分离,从而获得更快的酶反应动力学。此外,V254F突变引入了芳香残基苯丙氨酸,其芳环可与底物的π-电子体系之间形成π-π堆叠作用,增强了底物在催化构型下的结合,从而提高了酶的活性。
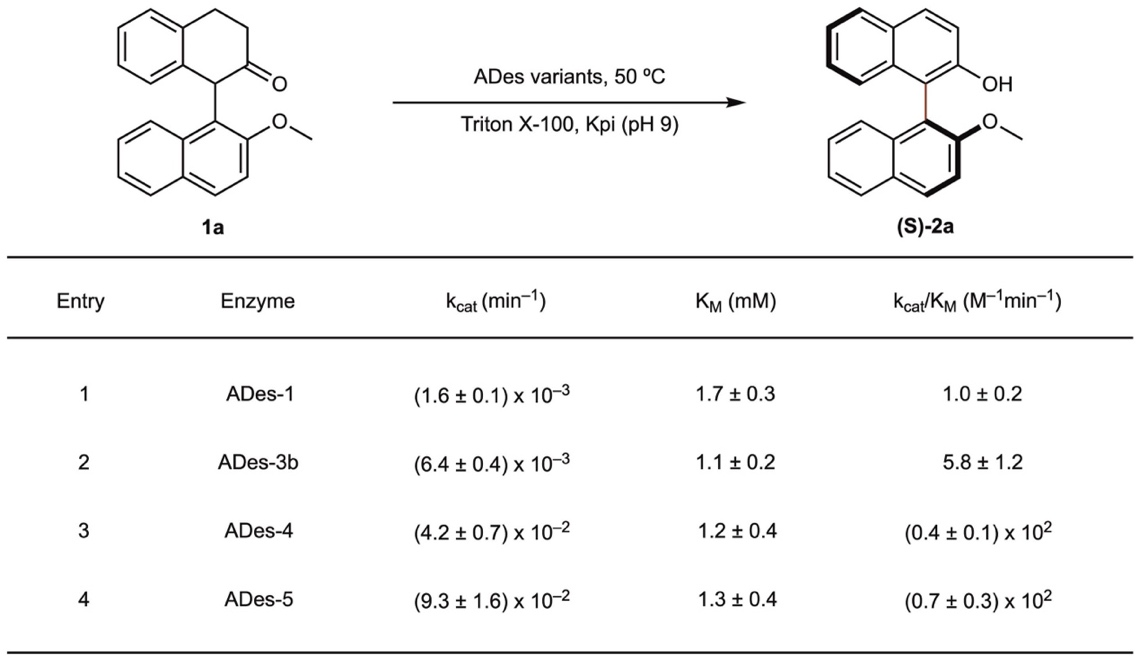
图3:ADes变体动力学参数的测定。
酶催化合成轴手性化合物的底物探索和合成应用
在确定最优变体ADes-5后,作者评估了其在多种1-芳基-2-四氢萘酮衍生物上的适用性。结果表明该酶对芳环的各种取代基都表现出显著的耐受性,无论是卤素、杂环还是氧化敏感的底物,均可被高效转化为目标联芳基产物,且具有良好的产率与对映选择性。除了1-萘基取代的四氢萘酮类化合物外,含有单芳环的底物也可以转化为相应的联芳基产物。值得注意的是,ADes-5在低温条件(4 °C)下对某些在常温下易发生消旋化的底物依然保持良好的去饱和活性,表明该酶在较宽的温度范围(4–50 °C)内仍具有良好的稳定性和催化能力。随后,实验人员成功将该酶催化反应放大至毫摩尔级别,所获得的联芳基产物中的酚羟基部分可通过 O-磺酰化和钯催化的交叉偶联反应,高效转化为多种功能化衍生物,包括芳基、硼基和膦基联芳基化合物,而且在这些偶联条件下没有观察到显著的消旋化,这进一步证明了该酶催化方法的实用性和目标产物的合成价值。
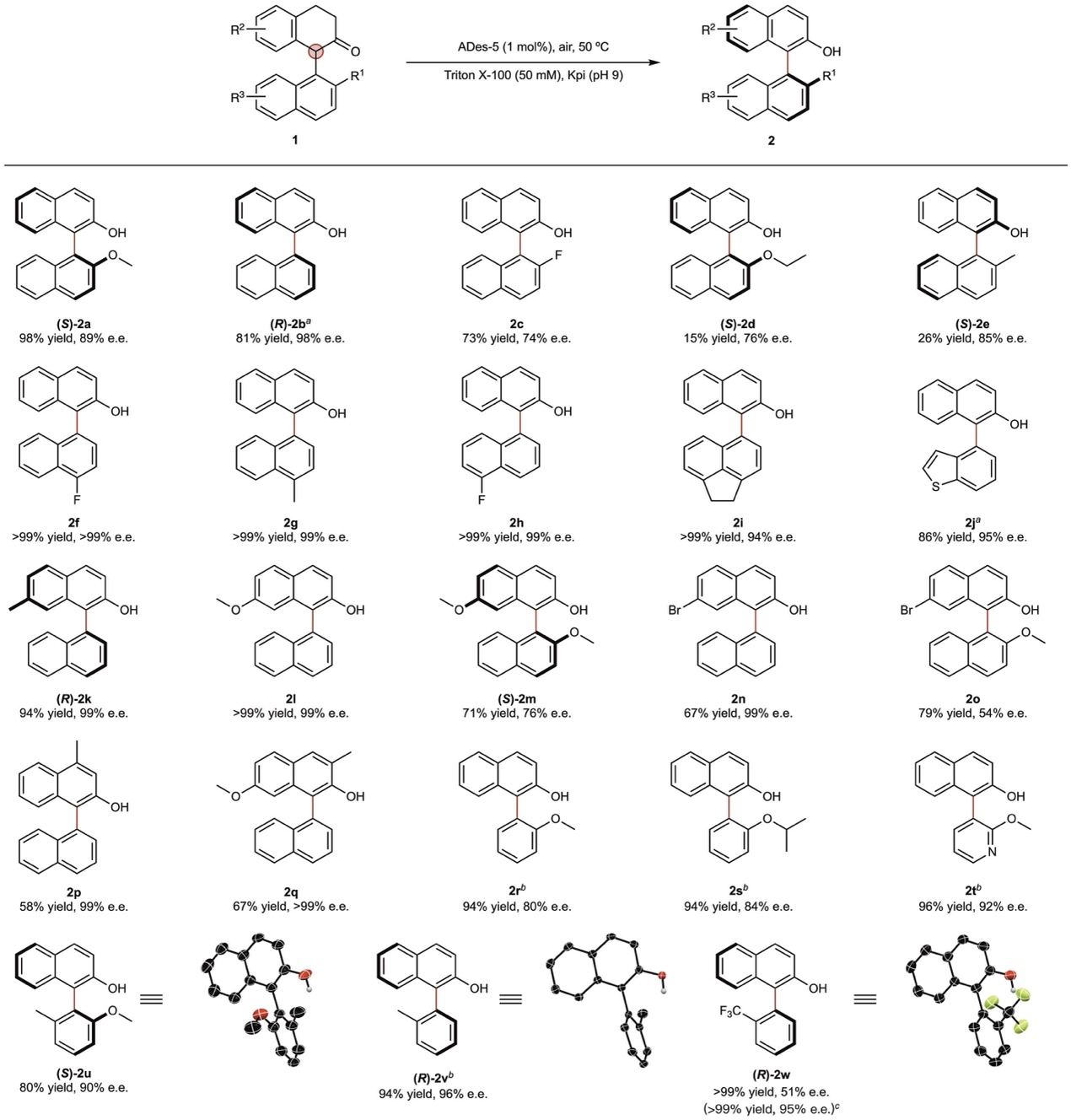
图4:酶催化反应底物的拓展。
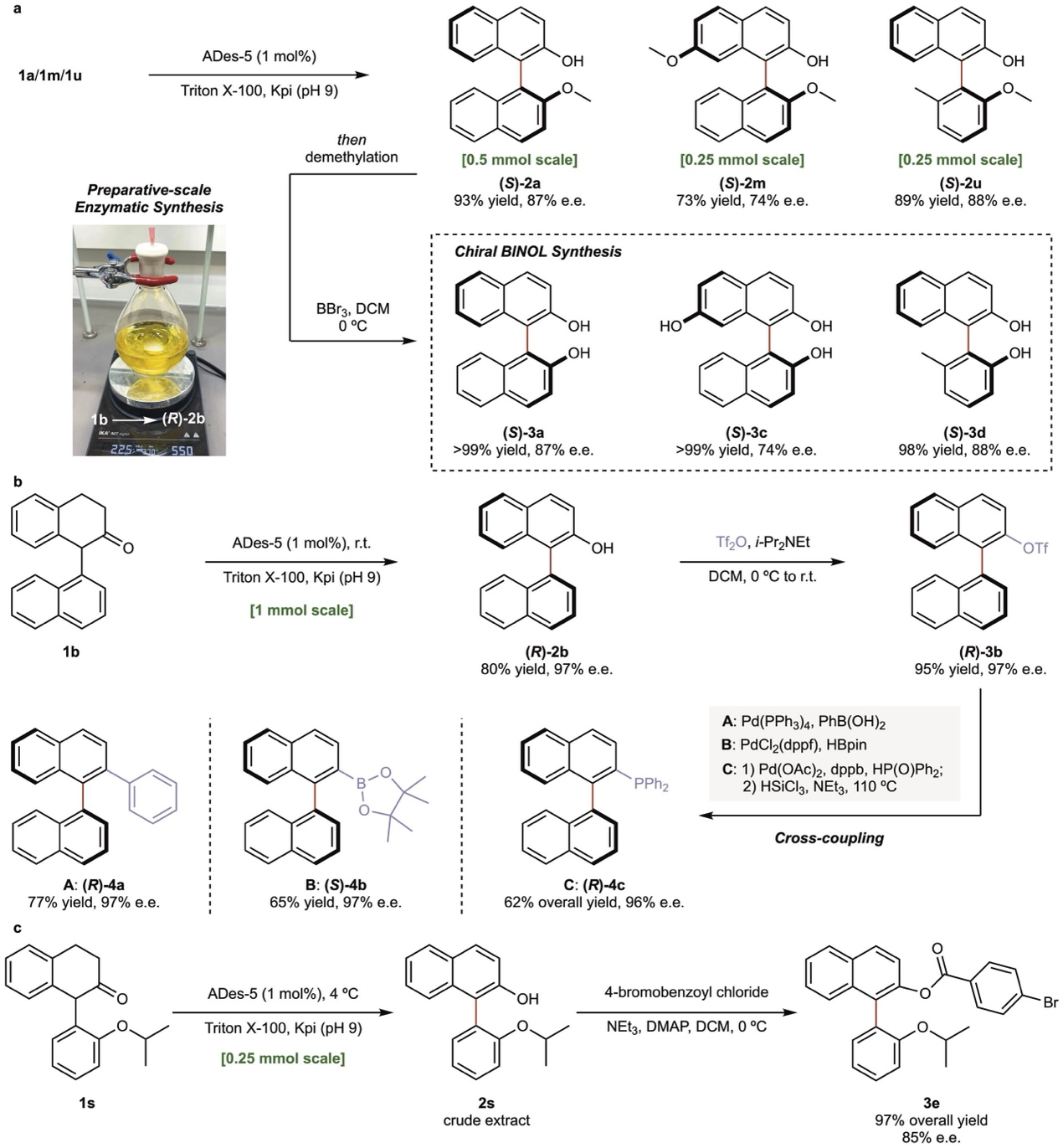
图5:制备级规模的放大和产物的衍生化。
综上所述,这项工作证明了黄素依赖的脱氢酶可用于轴手性分子的合成,扩大了这类酶的已知用途。同时,MD模拟有效预测了催化反应过程中酶的构象变化,并识别出以前在PtOYE和相关酶中未识别的重要残基,表明通过调控柔性loop的构象动力学可有效调节酶的催化活性,这一策略也为其他与生物催化转化相关的蛋白质体系的设计和优化提供了重要参考。
该项目得到科技部、清华大学生物医学交叉研究院以及北京生命科学研究所的资助。(来源:科学网)